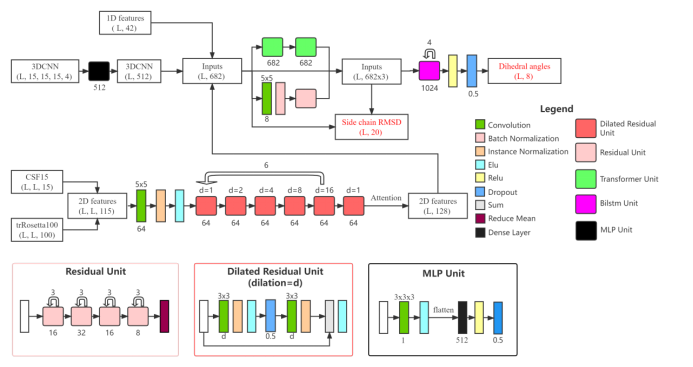
近日,浙江大学上海高等研究院计算+生物双聘教授、复旦大学复杂体系多尺度研究院院长马剑鹏及其团队提出了OPUS-Mut蛋白质侧链建模算法(图1),在进一步提升侧链建模准确率的同时,展示了该算法应用于蛋白质相互作用问题中的潜力。目前,这一成果已经开源,相关论文《Studying protein-protein interaction through side-chain modeling method OPUS-Mut》在线发表于Briefings in Bioinformatics。这项成果为新药研发提供了利器。
蛋白质三维结构由主链和侧链共同搭建而成,自然界中的蛋白质含有20种氨基酸,它们的主链几乎完全相同,而侧链差异很大。由于药物分子与人体蛋白质结合的位点绝大多数在氨基酸侧链上,如何精准的预测侧链位置对新药研发具有重要价值。这种精准预测能力还可用于解释基因点突变、基因小片段突变的机制,为遗传性疾病研究和治疗提供宝贵思路。
蛋白质侧链结构是蛋白质之间相互作用的基础,而蛋白质之间的相互作用又是多肽药物开发的关键。在蛋白质之间发生相互作用时,其位于接触面的残基的侧链结构起到了关键作用,发生了相应的构象变化。因此,精准地对接触面残基进行侧链建模是研究蛋白质之间相互作用的关键。
在对蛋白质多聚体的侧链建模中,尤其对位于接触面的氨基酸侧链建模中,OPUS-Mut展现了比其它侧链建模算法更高的准确率(图2、图3)。在蛋白质接触面上,预测更为准确的氨基酸侧链构象对研究蛋白质相互作用问题提供了帮助。
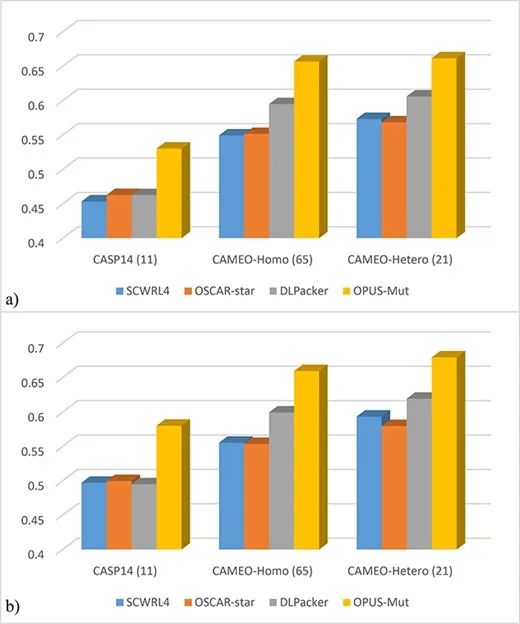
图2. 不同侧链建模算法在三个蛋白质多聚体测试集中的侧链建模性能
a)在全部氨基酸上的侧链建模准确率;b)在接触面氨基酸上的侧链建模准确率。
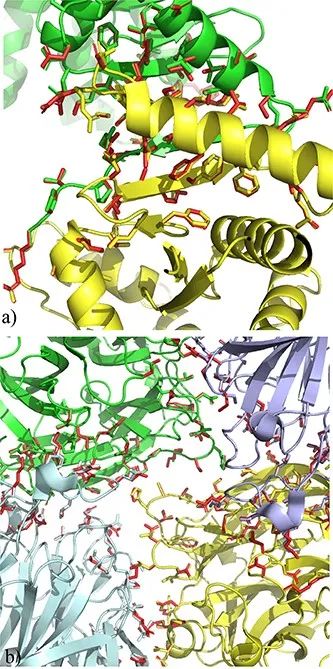
图3. OPUS-Mut侧链建模结果
绿色和黄色为多聚体中两条不同蛋白的天然构象,红色为OPUS-Mut的预测侧链结构。
在评价蛋白质对接构象时,预测更为准确的侧链构象是提升那些基于侧链位置信息的打分函数准确性的关键。值得注意的是,OPUS-Mut在预测残基侧链的同时,还提供其预测侧链的可信度。该可信度也可以代表该残基周围环境是否“舒适”。可信度越高,说明根据周围环境可以较容易的摆放该残基的侧链,其周围环境更接近训练集中的真实天然构象。
这种通过侧链对局部环境的反馈来对对接构象进行打分的方法,突出了侧链在对接中的重要性,为目前其它的打分函数提供了良好的补充,从而有利于对蛋白质对接构象的筛选和对其相互作用过程的模拟,这也是多肽药物开发中的重要问题。
其结果显示,基于OPUS-Mut的评分函数OPUS-Mut 相比于ZRANK,GNN-DOVE等评分函数可以更好的从虚拟对接构象集中区分出天然构象(图4)。
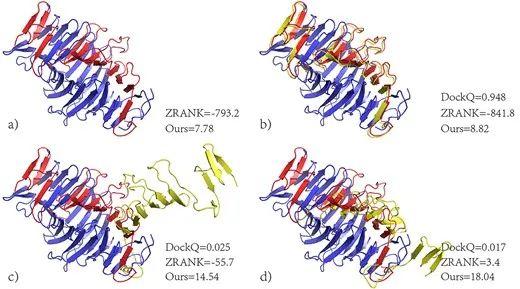
图4. 对蛋白质对接构象的评估结果
蓝色构象为受体蛋白的天然结构,红色构象为配体蛋白的天然结构,黄色构象为对接软件ZDOCK 3.0.2预测的排名前三的结果。“Ours”表示基于OPUS-Mut的评分函数OPUS-Mut。具有最低分数的构象被认为是天然构象。
马剑鹏团队在蛋白质侧链建模问题中深耕已久,相继开发了OPUS-Rota、OPUS-Rota2、OPUS-Rota3、OPUS-Rota4及OPUS-Mut等一系列算法,分别对应不同的应用场景,为多肽药物研发提供了支持。
文章可参见Studying protein–protein interaction through side-chain modeling method OPUS-Mut | Briefings in Bioinformatics | Oxford Academic (oup.com)
OPUS-Mut算法可通过http://github.com/OPUS-MaLab/opus_mut获取。
图文内容:转发自复旦大学复杂体系多尺度研究院
今日编辑:徐 敏


