生物大分子结构解析是基础生物科学的关键技术之一,冷冻电镜是其中非常重要的一种研究手段。
北京时间10月9日晚,复旦大学复杂体系度尺度研究院、浙江大学上海高等研究院双聘教授马剑鹏教授团队在先进智能算法上取得突破,开发新型计算方法OPUS-DSD,相关成果以“OPUS-DSD: deep structural disentanglement for cryo-EM single-particle analysis”为题,发表于Nature Methods(《自然-方法》)。
该算法不但能成功解析冷冻电子显微镜(Cryo-EM)结构解析技术中因传统方法无法分辨而缺损的生物大分子(比如蛋白质、核酸或蛋白质/核酸复合物等)结构,并且能高效精准地分辨出柔性结构域在受测样品中的构象分布。这一新方法能有效建立高精度的生物大分子结构模型,帮助解决药物设计中因目标蛋白结构不准而导致的新药研发失败问题。
研究人员介绍,生物大分子的许多重要功能是通过其高度的柔性特质来完成的,但柔性也是负面影响结构测定精度的主要因素。在处理冷冻电镜数据的过程中,生物大分子结构柔性引起的构象多样性使得从单个样本中获取精确的三维模型充满挑战,同时,由于冷冻电镜实验数据的信噪比通常极低,为深度学习算法在该领域的运用带来了巨大困难。
如何克服冷冻电镜数据中生物大分子结构的柔性、尤其是超大型复合物的柔性对结构测定精度带来的误差,是当前全球结构生物学研究的重点和难点,也是亟待打破的“瓶颈”。
该工作的智能算法成功地攻克了以上难题。研究团队推出了一种基于深度学习的计算方法,可有效地识别和处理生物大分子的柔性信息,从而提高冷冻电镜的解析能力,并获取三维结构的动态变化信息。图1与图2展示了两个典型结果。
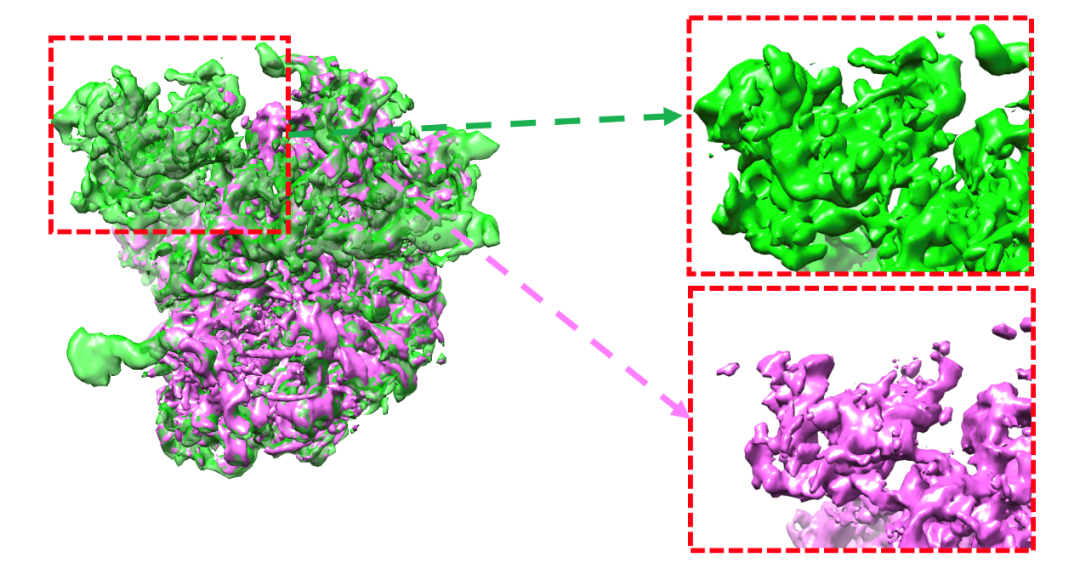
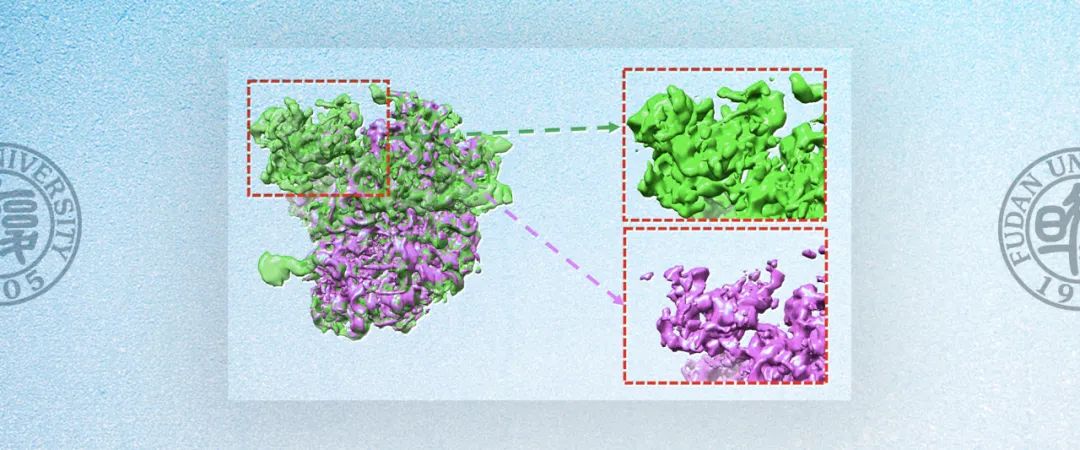
图1. OPUS-DSD重构结构模型与传统冷冻电镜软件解析的模型对比。在虚线标示的区域中,OPUS-DSD重构的模型(绿色)比传统冷冻电镜软件解析的模型(紫红色)有更加完整的电子密度。这是因为OPUS-DSD能分开重构不同三维构象,而不会将其重叠在同一个三维模型中。
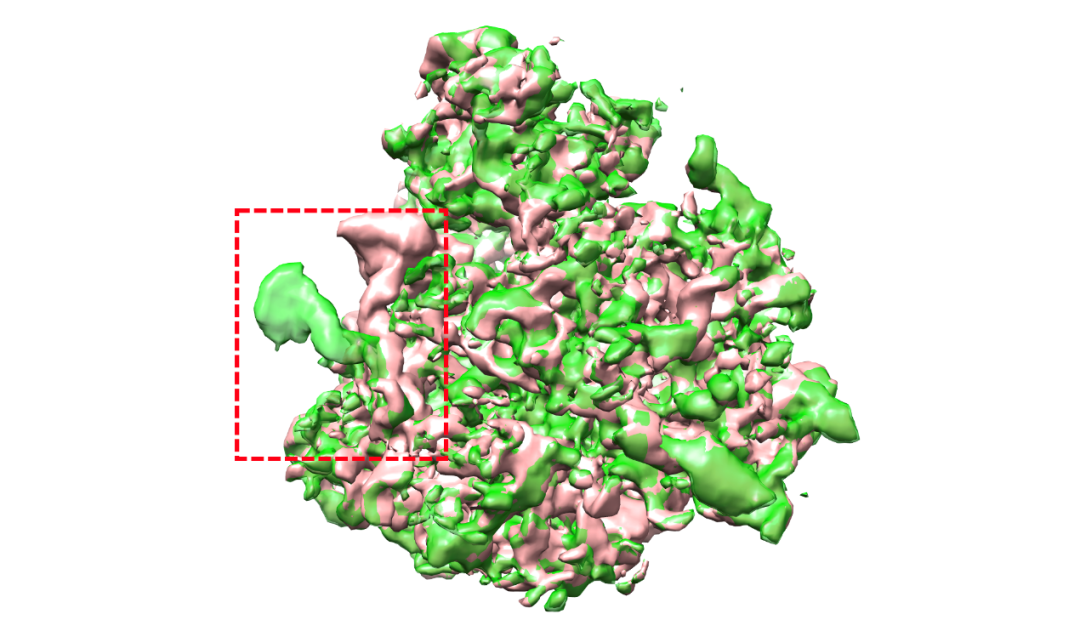
图2. OPUS-DSD解析的构象变化。绿色和古铜色分别代表OPUS-DSD解析的两个不同构象。在红色虚线框标识的区域,不同构象中的RNA链处在不同位置。这显示了该RNA链处在动态运动中。这种动态结构信息是很难用传统方法来提取和分辨的。
OPUS-DSD具有卓越的数据处理能力和鲁棒性,能在更低信噪比的数据上保持较高的解析准确性。此外,它不仅仅局限于单颗粒冷冻电镜技术,也可推广到更高端更低信噪比的冷冻断层扫描电镜(Cryo-ET)的研究中。
研究团队将继续以人工智能为技术中枢,构建新一代生物体系分析工具与方法,解读生物遗传信息,加速生命科学在分子层面的研究,通过对蛋白质、核酸等生物大分子功能结构的预测与设计,为药物研发提供支持,支撑全链条AI-赋能新药研发的先进技术平台。
算法在GitHub开源,地址为:https://github.com/alncat/opusDSD
论文链接:https://www.nature.com/articles/s41592-023-02031-6


